Estudio muestra cómo el microbioma gastrointestinal del camarón variaba entre organismos sanos e infectados

Una de las enfermedades más importantes que afecta a los países productores de camarón es la Necrosis Hepatopancreática Aguda (AHPND). La enfermedad es causada por bacterias del género Vibrio que albergan plásmidos [moléculas pequeñas de ADN extracromosómico dentro de una célula, separadas físicamente del ADN cromosómico y que pueden replicarse de forma independiente] pV-AHPND que codifican genes para toxinas. LA AHPND causa una alta mortalidad a través de un desprendimiento severo de las células hepatopancreáticas, lo que lleva a la muerte del camarón. Esta mortalidad conduce a pérdidas económicas considerables en la industria camaronera.
Existe una tendencia hacia la reducción del uso de antibióticos en la acuacultura, y el uso de probióticos – microorganismos que proporcionan efectos beneficiosos a sus huéspedes, ya sea previniendo la colonización de bacterias patógenas a través del antagonismo o promoviendo la salud animal mediante la estimulación del sistema inmunológico – es una alternativa prometedora y significativa a los antibióticos, y una forma de tratar y prevenir las enfermedades de los animales cultivados. Pero todavía existe la necesidad de identificar grupos beneficiosos de microorganismos entre la microbiota residente normal y los mecanismos subyacentes a las interacciones con los probióticos en el tracto gastrointestinal, para usarlos de manera eficiente para ayudar a mejorar la salud de los camarones cultivados.
La elección de los probióticos puede ser controvertida en algunos casos, particularmente en la acuacultura, donde ciertos géneros de probióticos están estrechamente relacionados filogenéticamente con los patógenos. Por ejemplo, en los criaderos de camarón ecuatorianos, las cepas beneficiosas pertenecientes a la especie de bacteria Vibrio alginolyticus y las cepas causantes de AHPND de la especie Vibrio parahaemolyticus pertenecen al mismo clado filogenético [un grupo de organismos compuestos por un ancestro común y todos sus descendientes lineales], el clado Harveyi, y se han aislado de cultivos de larvas sanas y no sanas. En particular, una cepa anteriormente conocida como V. alginolyticus ILI [las herramientas genómicas actuales y los genomas disponibles nos permitieron clasificarlos como Vibrio diabolicus] se ha aislado de larviculturas de camarones sanos y se ha demostrado que es un agente probiótico eficaz.
Este artículo – adaptado y resumido de la publicación original [Restrepo et al. (2021). Microbial community characterization of shrimp survivors to AHPND challenge test treated with an effective shrimp probiotic (Vibrio diabolicus). Microbiome 9, 88 (2021) – reporta sobre un estudio para evaluar el efecto de la cepa ILI y su potencial para limitar los efectos perjudiciales de AHNPD utilizando metabarcoding [el código de barras de ADN / ARN de una manera que permite la identificación simultánea de muchos taxones dentro de la misma muestra] en la microbiota gastrointestinal de camarones después de haber sido desafiados con V. parahaemolyticus causante de AHPND.
Configuración del estudio
Se utilizó la cepa probiótica ILI proporcionada por el Laboratorio de Microbiología del Centro Nacional de Acuicultura e Investigaciones Marinas (CENAIM), previamente identificada como V. alginolyticus y caracterizada como un probiótico eficiente. Esta cepa se aisló originalmente del agua de un criadero de larvas de camarones sanos y ha sido parte de la colección de cultivos del CENAIM durante más de 26 años.
Después de alimentar al camarón blanco del Pacífico (Litopenaeus vannamei) con los correspondientes tratamientos probióticos, se evaluó la supervivencia de los animales a un desafío con la cepa V. parahaemolyticus BA94C2.
Se aplicó un diseño completamente al azar, con cuatro tratamientos y seis repeticiones por tratamiento. Las unidades experimentales (tanques de 40 litros) se sembraron con 40 camarones (2.5 ± 0.3 gramos). Durante el ensayo, se controló la mortalidad de los camarones cada dos horas. Se analizaron las diferencias de mortalidad acumulada 50 horas después de la infección entre los tratamientos infectados y se registró la supervivencia de los camarones al final del experimento. El intercambio de agua al 50 por ciento diario se realizó antes y después del desafío, incluso durante el período de alimentación con probióticos, pero no durante el período de desafío de 50 horas. Los camarones se expusieron a un fotoperiodo de 12 horas por día.
Para obtener información detallada sobre la preparación del inóculo probiótico, la prueba de desafío por inmersión de camarones suplementados con probióticos y las diversas pruebas realizadas, consulte la publicación original.
Resultados y discusión
Evaluamos los cambios en la estructura de la comunidad microbiana causados por AHPND en estómagos de camarón y hepatopáncreas, con y sin presencia de probióticos. Los camarones no fueron desafiados (controles sanos; C0) o desafiados con la cepa de V. parahaemolyticus BA94C2 causante de AHPND (controles infectados con AHPND; C1) y se compararon con la misma configuración, pero con la suplementación diaria de un probiótico (proporcionado en el alimento comercial y sin otro medicamento probiótico) durante un período de un mes antes del desafío. Usamos como probiótico la cepa ILI (C2) y otro probiótico (Bacillus sp. Cepa P64) para la comparación (C3). Los resultados de la prueba de desafío mostraron los valores de supervivencia más altos para los tratamientos C0 y C2 seguidos de C3. Los camarones experimentales con mayor mortalidad fueron los del tratamiento de camarones infectados con AHPND (C1).
Hasta la fecha, hay información muy limitada sobre el efecto de AHPND en el microbioma del camarón [comunidad microbiana característica que ocupa un hábitat razonablemente bien definido que tiene propiedades fisicoquímicas distintas], y los estudios existentes se han centrado en la microbiota intestinal. Si bien no es clara la contribución de la microbiota intestinal al impacto de la enfermedad AHPND, destacamos que la diversidad en el intestino es mayor que en el hepatopáncreas pero similar a la que se encuentra en el sedimento.
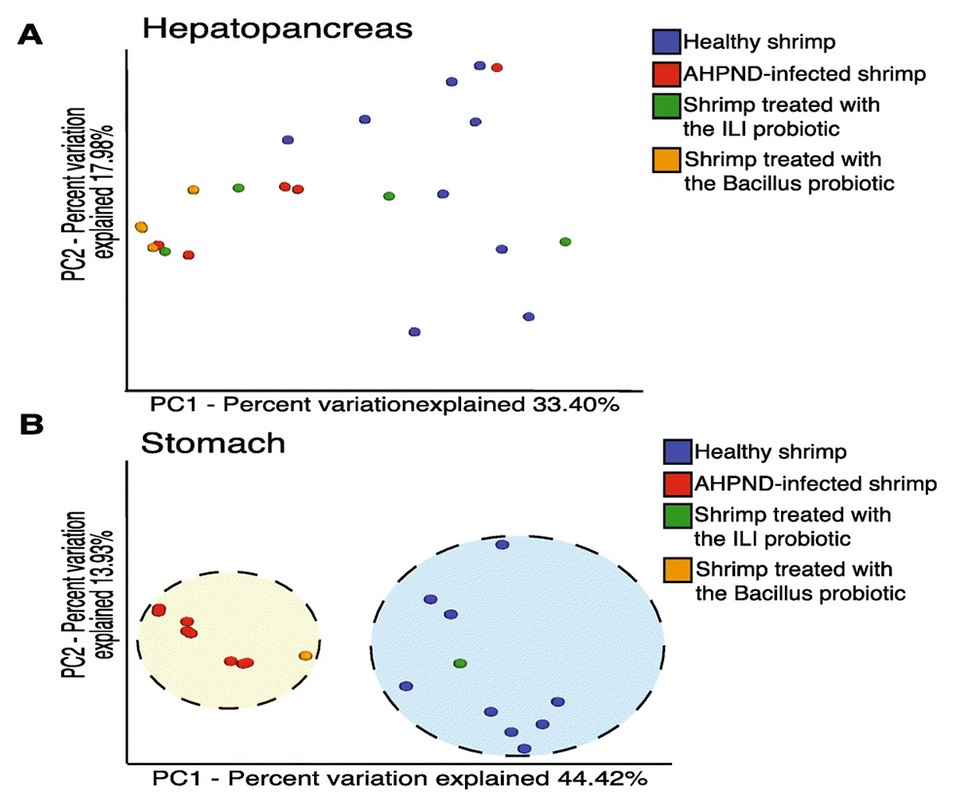
Se sabe que los órganos relacionados con el sistema inmunitario – como los hemocitos, el hepatopáncreas y las branquias – son importantes en la respuesta a la enfermedad de AHPND. Además, la colonización del estómago pareció ser el paso inicial de la infección por Vibrio causante de AHPND. Además, AHPND afecta principalmente al hepatopáncreas y el estómago de los camarones infectados; sin embargo, caracterizar la microbiota del estómago de los camarones no ha sido el enfoque principal para comprender los cambios en las respuestas de la microbiota. Por lo tanto, examinamos las alteraciones del microbioma en respuesta a las bacterias patógenas que producen AHPND, en el estómago y el hepatopáncreas de los camarones.
Identificamos una pérdida significativa de diversidad en la microbiota del hepatopáncreas y el estómago asociada con AHPND. Nuestros resultados contrastan con algunos efectos mínimos observados en la microbiota intestinal de los camarones después de la infección por el Virus del Síndrome de la Mancha Blanca (WSSV) y AHPND. Así, consideramos que la abundancia y composición de la microbiota se ven influidas en función del órgano en el que se ubica.
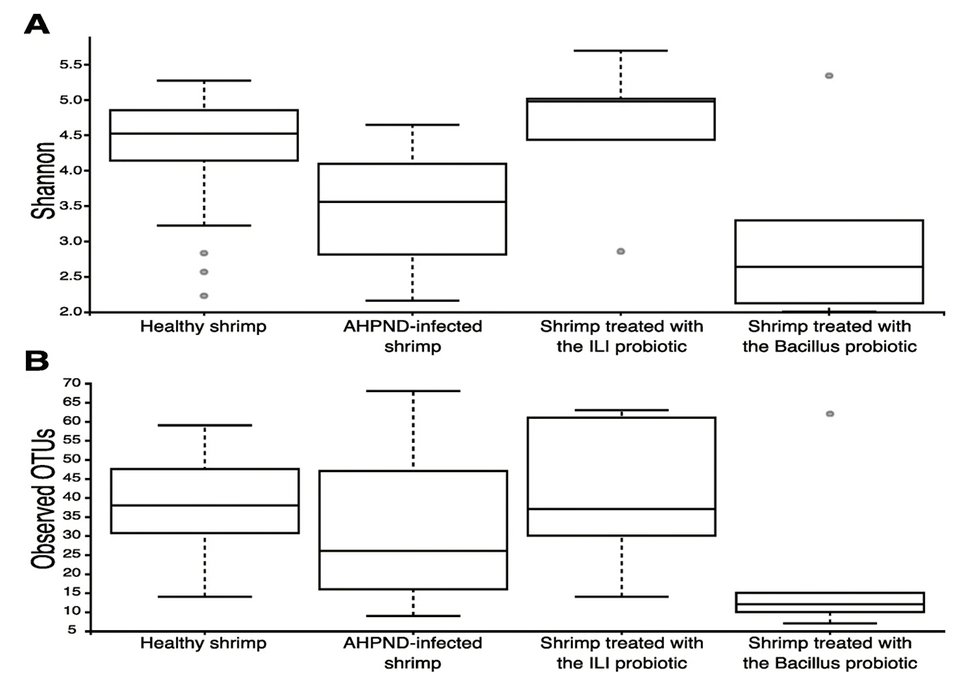
Fue interesante que identificamos miembros del filo Tenericutes, en particular los asignados al género Mycoplasmatales, como miembros del microbioma sano. Cuatro variantes de secuencia de amplicones, ASV [que se refieren a secuencias de ADN únicas recuperadas de un análisis de genes marcadores de alto rendimiento] de Mycoplasmatales pertenecían al género Mycoplasma, siendo el grupo taxonómico más abundante del microbioma del estómago sano.
Varios estudios sobre el papel de Mycoplasma spp. en otros organismos han sugerido que serían comensales inofensivos [microorganismos que viven continuamente en, o en ciertas partes del cuerpo, sin causar enfermedad] o endosimbiontes [cualquier organismo que viva dentro del cuerpo o células de otro organismo con mayor frecuencia, aunque no siempre, en una relación mutualista] con el anfitrión; sin embargo, se desconoce su papel específico dentro del microbioma del camarón. Se han asociado cepas específicas de Mycoplasma con camarones sanos, lo que sugiere potencialmente que tiene una función en la salud del huésped y en la recuperación de estados patológicos.
Pudimos observar que el uso de probióticos bacterianos como ILI puede modular la microbiota del huésped y podría contribuir como estimulante para varias especies que tienen potencial probiótico al inhibir la colonización gastrointestinal de patógenos, como encontramos en este estudio.
El hecho de que ILI pueda adquirir el plásmido causante de AHPND muestra la alta capacidad de las especies de Vibrio para la transferencia horizontal de genes, HGT [el movimiento de material genético entre organismos unicelulares y / o multicelulares que no sea por la transmisión (“vertical”) de ADN desde progenitores a hijos (reproducción)], que puede utilizarse para ocupar diferentes nichos. La adquisición puede variar en el medio acuático y puede estar influenciada por los antecedentes genéticos. El mantenimiento de estas características en bacterias no patógenas podría ser una prueba de que las toxinas secretadas son características evolutivamente antiguas que pueden desempeñar un papel más importante en la aptitud ambiental.
El plásmido ILI no se ha descrito en ningún informe anterior, pero podría proporcionar funciones importantes en las capacidades probióticas que exhibe esta cepa. Es intrigante que ninguna otra cepa patógena de Vibrio reportada contenga un plásmido con contenido genético similar. La mayoría de los plásmidos grandes que están asociados con especies de Vibrio se han caracterizado por ser altamente virulentos contra diferentes hospedadores. Curiosamente, no encontramos ningún factor de virulencia potencial codificado en el plásmido ILI; por tanto, el papel del plásmido en las bacterias aún no está claro. Más estudios sobre el papel del plásmido en la cepa de ILI podrían aclarar su función y, potencialmente, arrojar luz sobre si contribuye a las capacidades probióticas de ILI.
En conjunto, nuestros datos indican que la virulencia es multifactorial en el clado Harveyi, y que las fuentes de virulencia podrían estar asociadas a huéspedes específicos ya que muchos de los genes asociados pueden estar involucrados en la supervivencia de las bacterias en diferentes ambientes.
Perspectivas
El uso de probióticos en la acuacultura es una alternativa práctica para promover la salud animal y prevenir enfermedades. Caracterizamos las alteraciones del microbioma generadas por un probiótico exitoso que podría controlar las poblaciones patógenas en el tracto gastrointestinal del camarón y estimular la supervivencia en la acuacultura.
La cepa de V. diabolicus ILI es una cepa bacteriana aislada del medio ambiente en un cultivo de larvas de camarón que ha mostrado actividad antimicrobiana contra muchas cepas patógenas de diferentes especies de Vibrio. La suplementación de ILI a L. vannamei no solo disminuyó la abundancia de variantes de secuencia de amplicones (ASV) de Proteobacteria, sino que también mantuvo una comunidad microbiana similar a la saludable en el tracto gastrointestinal del camarón después de ser desafiado con bacterias causantes de AHPND.
Nuestros hallazgos sugieren que la cepa ILI probablemente se puede usar como probiótico para reducir la población de patógenos para AHPND en el camarón y para mejorar la supervivencia y la resistencia contra esta importante enfermedad en la acuacultura del camarón, sin el riesgo de convertirse en un organismo patógeno.
Now that you've finished reading the article ...
… we hope you’ll consider supporting our mission to document the evolution of the global aquaculture industry and share our vast network of contributors’ expansive knowledge every week.
By becoming a Global Seafood Alliance member, you’re ensuring that all of the pre-competitive work we do through member benefits, resources and events can continue. Individual membership costs just $50 a year. GSA individual and corporate members receive complimentary access to a series of GOAL virtual events beginning in April. Join now.
Not a GSA member? Join us.
Authors
-
Leda Restrepo, M.Sc.
Department of Biological Sciences, Universidad de los Andes, Bogotá, Colombia; Max Planck Tandem Group in Computational Biology, Universidad de los Andes, Bogotá, Colombia; Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador
-
Cristóbal Domínguez-Borbor, M.Sc.
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador
-
Leandro Bajaña
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador -
Irma Betancourt
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador
-
Jenny Rodríguez, Ph.D.
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador; Escuela Superior Politécnica del Litoral, ESPOL, Facultad de Ciencias de la Vida, FCV, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador
-
Bonny Bayot, Ph.D.
Escuela Superior Politécnica del Litoral, ESPOL, Centro Nacional de Acuicultura e Investigaciones Marinas, CENAIM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador; Escuela Superior Politécnica del Litoral, ESPOL, Facultad de Ingeniería Marítima y Ciencias del Mar, FIMCM, Campus Gustavo Galindo Km 30.5 Vía Perimetral, P.O. Box 09-01-5863, Guayaquil, Ecuador
-
Alejandro Reyes, Ph.D.
Corresponding author
Department of Biological Sciences, Universidad de los Andes, Bogotá, Colombia; Max Planck Tandem Group in Computational Biology, Universidad de los Andes, Bogotá, Colombia; Center for Genome Sciences and Systems Biology, Department of Pathology and Immunology, Washington University in Saint Louis, Saint Louis, MO, USA


